シングルセル遺伝子発現解析とは
シングルセル遺伝子発現解析とは、従来のように細胞集団(バルク)として平均的に遺伝子発現を解析する手法とは異なり、単離した1細胞ごとに遺伝子発現を解析する手法です。これにより、同一組織や細胞集団の中に存在する細胞の多様性や不均一性を詳細に明らかにすることが可能となります。
従来のバルク解析では捉えることが難しかった、がん組織における多様な細胞集団や免疫抑制環境なども、シングルセル遺伝子発現解析の導入により徐々に解明されつつあり、がん治療や免疫療法への応用が期待されています。
シングルセル解析は、細胞集団の不均一性を高解像度で捉える手法として研究分野で急速に普及し、現在では多様な解析プラットフォームが開発されています。各プラットフォームにはそれぞれ特長や制約があるため、研究目的やサンプル特性、予算(装置導入コストおよびランニングコスト)に応じて最適な手法を選択することが重要です。
解析手法やプラットフォームによって詳細な工程には違いがありますが、シングルセル遺伝子発現解析の基本的なプロセスは共通しており、以下にその概要を示します。
シングルセル遺伝子発現解析フローチャート
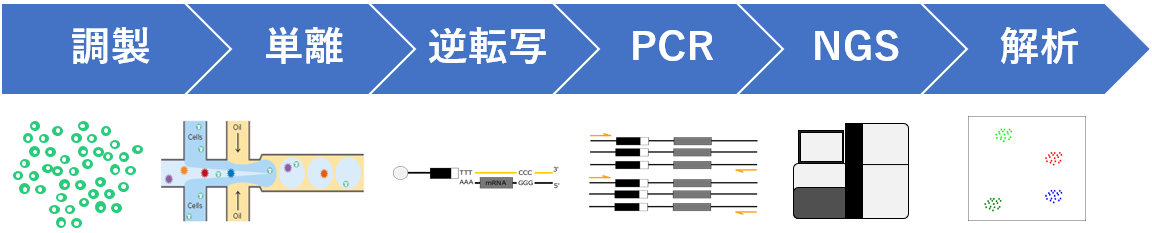
細胞・核などのサンプル懸濁液調製
シングルセル解析では、まず解析対象となる組織や試料から、シングルセル(単一細胞)または単一核のサンプル懸濁液を調製します。この工程では、組織の種類や性状に応じて、酵素処理や機械的分散などを用いて細胞を分離し、個々の細胞(または核)が均一に分散した状態を作成します。
調製対象となるサンプルには、組織、血液、生検体、腫瘍、培養細胞をはじめ、植物片、真菌類(酵母など)、プロトプラストなど、多様な生物由来試料が含まれます。また、組織の保存状態や細胞の脆弱性によっては、単一核(snRNA-seq)を用いた解析が選択される場合もあります。
このサンプル懸濁液調製工程は、後続の単離、mRNA捕捉、遺伝子発現解析の品質に大きく影響するため、細胞の生存率、凝集の有無、不純物の除去などに注意しながら、適切な条件で行うことが重要です。
»» 各種調製方式
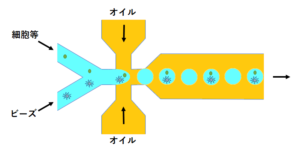
単離・mRNA捕捉(ドロップレット式の場合)
シングルセル(1細胞)を、ドロップレットやウェルなどの微小区画に分配し、個別バーコード(識別)配列を有するmRNA捕捉粒子と1細胞ずつペアリングして単離します。
ドロップレット方式では、これらの微小区画はドロップレットジェネレーターによって生成され、細胞懸濁液とmRNA捕捉粒子が制御された流体条件下で効率よく封入されます。
この区画化の過程では、1つの区画内に2個以上の細胞が同時に入る確率(ダブレット率)が重要な指標となります。ダブレットが発生すると、複数細胞由来の遺伝子発現情報が同一細胞のデータとして混在して検出されるため、細胞集団の誤分類や解析結果の解釈に影響を与える可能性があります。
そのため、シングルセル解析プラットフォームを選定する際には、ダブレット率を性能評価の重要な指標として比較することが不可欠です。また、投入する細胞数が増加すると統計的にダブレット率も上昇するため、異なるプラットフォームを比較する際には同一投入細胞数条件でのダブレット率を確認することが重要です。
細胞の単離時には、細胞溶解液とmRNA捕捉粒子をあらかじめ同一溶液として用意し、ドロップレット生成の過程で細胞とともに封入します。これにより、細胞はドロップレット内で初めて細胞溶解液およびmRNA捕捉粒子と接触し、区画形成と同時に細胞溶解およびmRNA捕捉が開始されます。
この設計により、細胞単離後の操作を最小限に抑えつつ、細胞間の混入を防ぎながら、再現性の高いシングルセル遺伝子発現解析を実現します。
逆転写・cDNA合成
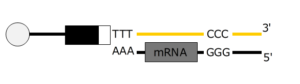
バーコード(識別)配列を持ったビーズやハイドロゲルなどのmRNA捕捉粒子によって捕捉されたmRNAは、ドロップレットやウェルから回収されます。これらのmRNAには、どの細胞由来であるかを示す細胞バーコードや、個々のmRNA分子を区別するためのユニーク分子識別子(UMI)が付加されています。
回収されたmRNAを鋳型として、逆転写酵素を用いた逆転写反応を行うことで、mRNA配列に対応するcDNA(相補的DNA)の第1鎖が合成されます。この際、mRNAに付加されていたバーコード配列やUMI情報も同時にcDNAへと引き継がれるため、後の解析において、どの細胞からどの遺伝子が発現していたかを識別することが可能になります。
この逆転写/cDNA合成工程は、RNAという不安定な分子情報を、安定で増幅可能なDNA情報へ変換する重要なステップであり、以降のライブラリー調製や次世代シーケンシング解析の基盤となります。
PCR増幅・品質評価・ライブラリー調製
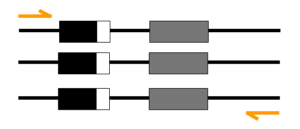
逆転写反応によって合成されたcDNAを鋳型として、特定の配列に結合するプライマーを用い、PCR(Polymerase Chain Reaction)によりcDNAを増幅します。この工程により、次世代シーケンシング(NGS)に十分な量のDNAを得ることが可能になります。
PCR後には、得られた増幅産物を回収し、電気泳動やフラグメント解析などを用いてサイズ分布や増幅状態を確認します。これらの品質チェックにより、非特異的な増幅や過度な断片化が起きていないかを評価し、解析に適したサンプルであるかを判断します。
品質が確認されたPCR産物は、NGS用キットを用いてライブラリー調製を行います。この工程では、シーケンシングに必要なアダプター配列やサンプル識別のためのインデックス配列が付加され、NGS装置で直接解析可能な状態に変換されます。
NGS解析はコストや解析リソースを要するため、品質が十分に確保されたサンプルのみを次工程へ進めることが重要です。事前に適切な品質評価を行うことで、無駄な解析を防ぎ、信頼性の高いシングルセル遺伝子発現データを取得することができます。
NGS(次世代シーケンシング)

ライブラリー調製されたDNAを用いて、Illumina社などの次世代シーケンサーによる配列解析を行います。シングルセルRNA解析では、主にNextSeq 1000/2000やNovaSeq Xシリーズなどのプラットフォームが用いられ、解析規模や目的に応じて機種を選択します。
シーケンシングによって得られたリード配列には、細胞バーコードおよびUMI情報が含まれており、これらをもとに 細胞ごとの遺伝子発現情報を高精度に再構築することが可能です。NGS工程は最終的なデータ品質を左右する重要なステップであり、目的に応じた適切なプラットフォーム選択が求められます。
データ解析
シングルセル解析では、NGSによって得られたシーケンスデータに対して、複数の解析ステップを段階的に実行します。
まず、Cell Ranger などの一次解析ソフトを用いて、リード配列のトリミング、参照ゲノムへのマッピング、細胞バーコードおよびUMI情報に基づく発現量の定量を行い、細胞×遺伝子の発現行列を作成します。
得られた発現データは、Seurat(R)やScanpy(Python)といった二次解析ソフトを用いて解析されます。これらのツールでは、品質管理(QC)、正規化、バッチ効果補正などを行い、細胞間の技術的ばらつきを低減します。
その後、次元削減(PCA、UMAPなど)やクラスタリング解析を行うことで、遺伝子発現パターンが類似した細胞群を同定します。最終的に、マーカー遺伝子の発現解析や可視化を通じて、細胞タイプの推定や状態変化の解析など、生物学的な解釈につなげていきます。
データ解析結果の例(UMAP図・t-SNE図・クラスタリング)
データ解析の結果は、UMAP(Uniform Manifold Approximation and Projection)やt-SNE(t-Distributed Stochastic Neighbor Embedding)などの次元削減手法を用いて可視化されることが一般的です。これらの図では、各点が1細胞を表し、遺伝子発現パターンが類似した細胞同士は近くに配置されます。
UMAPは、全体構造と局所構造のバランスを保ちつつ可視化でき、再現性や大規模データへの適用性に優れるため、近年のシングルセル解析で広く用いられています。
一方、t-SNEは局所的な類似性の強調に優れており、近い細胞同士の関係性を直感的に把握するのに適しています(全体構造の解釈には注意が必要です)。
これらの可視化上で形成される集団(クラスタ)は、共通した遺伝子発現特徴を持つ細胞群を示します。クラスタごとにマーカー遺伝子の発現を確認することで、細胞タイプや細胞状態の推定を行うことができます。このような解析結果を通じて、条件依存的な変化や新規細胞サブタイプの発見につなげることが可能です。
シングルセルRNA-seq解析及びシングルセルレパトア解析の受託サービス
受託サービスは下記にて提供されております。
- 株式会社CyberomiX(外部ページへ)
- KOTAIバイオテクノロジーズ株式会社(外部ページへ)
- 大阪大学微生物病研究所 遺伝情報実験センター(外部ページへ)
参考文献
- Gross, Andre et al. “Technologies for Single-Cell Isolation.” International journal of molecular sciences vol. 16,8 16897-919. 24 Jul. 2015, doi:10.3390/ijms160816897
- Macosko, Evan Z et al. “Highly Parallel Genome-wide Expression Profiling of Individual Cells Using Nanoliter Droplets.” Cell vol. 161,5 (2015): 1202-1214. doi:10.1016/j.cell.2015.05.002
- Nguyen, Quy H et al. “Experimental Considerations for Single-Cell RNA Sequencing Approaches.” Frontiers in cell and developmental biology vol. 6 108. 4 Sep. 2018, doi:10.3389/fcell.2018.00108
- Valdes-Mora, Fatima et al. “Single-Cell Transcriptomics in Cancer Immunobiology: The Future of Precision Oncology.” Frontiers in immunology vol. 9 2582. 12 Nov. 2018, doi:10.3389/fimmu.2018.02582
- Valihrach, Lukas et al. “Platforms for Single-Cell Collection and Analysis.” International journal of molecular sciences vol. 19,3 807. 11 Mar. 2018, doi:10.3390/ijms19030807